Machine Learning in der Google Cloud 2

Prinzipiell ist gegen Random Forests nichts einzuwenden, wenn man auf Retro-Modelle aus den 90ern steht. Manche sagen auch “bewährte Technik”. Allerdings muss man es sich auch erst einmal leisten können, die entsprechende Hardware anzuschaffen. Denn es ist das Eine, die Modelle zum Laufen zu bringen, aber das Andere, dass sie zu Lebzeiten auch fertig werden. Man freut sich ja, wenn der übergequollene Arbeitsspeicher das Tuning nicht im Keim erstickt, weiß aber den schnellen Tod zu schätzen, wenn man gespannt auf Ergebnisse wartet, die nie geliefert werden.
Wogegen aber schon etwas einzuwenden ist, ist die sehr niedrige Meme Dichte im letzten Blog-Beitrag! Die Dichte verkam eher zum Vakuum. Viele Menschen haben mich auf der Straße angesprochen, ob der überbordenden Seriosität, für die ich mich an dieser Stelle entschuldigen möchte. Dieser Beitrag soll sich als Fels in der Brandung erheben, der sich der stroke-Betroffenheitswelle entgegenstemmt, die danach trachtete, jeglichen humoristischen Ansatz im Keim zu ersticken.
Also, worum wird es im zweiten Teil gehen? Wir schauen uns mlr3pipelines etwas detaillierter an. Mit Pipelines kann man wunderbare Dinge machen, wie verschiedene Modelle parallel laufen zu lassen bzw. sie hintereinanderzuschalten (stacking). Abbildung 1 zeigt uns gleich mal eine etwas komplexere Pipeline, wie sie auf unser stroke Klassifikationsproblem angewendet wurde. Schön bunt. Also was passiert hier?
- Im ersten Branch können wir uns nicht entscheiden, ob wir die Kategorie
Unknownin der Variablesmoking statusimputieren sollen (nach dem Motto: irgendeinen wahrensmoking statusmüssen die Leute ja haben) oder ob wir einfach die Kategorie so lassen (nach dem Motto: Leute von denen man es nicht weiß, sind qualitativ eventuell anders als Leute, von denen man den Status weiß). Diese Frage eignet sich hervorragen, um an Teilnehmer:innen in einem Statistik gerichtet zu werden. Nach 20 Minuten Diskussion, lernen alle, dass es nicht immer eine richtige Antwort gibt. - Wir können uns nicht für ein Modell entscheiden. Daher trainieren wir mal mehrere Modelle parallel und leiten die Ergebnisse dann an unseren Super Learner (
xgboost) weiter. Der soll dann das beste draus machen. Und Achtung:xgboostist nicht nur Super, sondern per Definition schon extrem.
Ich wollte eigentlich recht ausführlich über die Ergebnisse berichten und dieses kompliziertere Modell mit dem einfachen Random Forest vergleichen. Aber gut, wayne interessierts? Also stürzen wir uns auf die Pipe!
Warum Pipelines?

Jedem Anfang wohnt ein gewisser Zauber inne, sagen die einen. Die anderen sagen, dass man lieber die Finger von was Neuem lassen sollte, denn es kostet Zeit, Nerven und am Ende bleibt man, mit einer hohen Wahrscheinlichkeit, doch bei dem, was man vorher schon gekannt hat. Der bekannte Horror wird bevorzugt. Mir geht es mit neuen Packages meistens so, dass der Zauber ihnen nur so lange innewohnt, solange ich mich oberflächlich mit dem Thema beschäftigt habe, Stichwort: Vorträge und YouTube-Videos. mlr3 wirkt wie die Lösung aller Machine-Learning Probleme, bis zu dem Zeitpunkt, bei dem es sich dann selbst in die Schlange dieser Probleme einreiht. Nach dem Motto: Bevor ich mlr3 probiert habe, hatte ich ein Problem beim Tunen meines Modells, jetzt habe ich zwei. Tatsächlich (auch nachhaltig) positiv in diesem ganzen Prozess sind mir die mlr3pipelines aufgefallen. Mit einer Pipeline kann man quasi ein Rezept erstellen, das beschreibt, was mit meinen Daten alles passieren soll, ohne dass man selbst kochen muss.
Die Aufarbeitungsschritte werden nicht sofort umgesetzt, wie z. B. Variablen imputieren oder standardisieren, sondern die Daten werden erst im Laufe des Tuning Prozesses, immer wieder, aufbereitet. Manchmal ist die Aufbereitung (wie bei uns) auch Teil des Modells. Insbesondere verhindert man, z. B. im Falle einer Crossvalidation (CV), dass Infos von aus dem Trainingsdatensatz in den Test Datensatz “leaken”. Auch hilfreich ist, dass man bestimmte Entscheidungen vielleicht nicht durch “Expertenwissen” treffen will1, sondern man kann auch einfach schauen was den besseren Fit ergibt. Probieren geht über studieren. Außerdem, kann man dann coole Grafiken machen.
Pipeline mit dem stroke Datensatz
Hier ist der Code, der die Pipeline beinhaltet die in Abbildung 1 dargestellt ist.
Code: Stacking mit mlr3 (Achtung: Viel Code!)
library(data.table)
library(magrittr)
library(mlr3)
library(mlr3tuning)
library(mlr3extralearners)
library(mlr3learners)
library(mlr3pipelines)
library(paradox)
library(bbotk)
library(mlr3mbo)
library(forcats)
library(future)
library(xgboost)
plan(multisession)
## Argumente uebernehmen
args <- commandArgs(trailingOnly = TRUE)
# defaults
default_evals <- 10
default_duration <- 1 * 60 * 60
n_evals <- ifelse(length(args) >= 1, as.integer(args[1]), default_evals)
duration <- ifelse(length(args) >= 2, as.integer(args[2]), default_duration)
cat("\n -------------------------------------------- \n")
cat("Tuning Stoppt nach:\n")
cat(" Evals:", n_evals, "\n")
cat(" Zeit in Sekunden:", duration)
cat("\n -------------------------------------------- \n\n")
set.seed(42)
# einlesen und modden
d = fread("healthcare-dataset-stroke-data.csv") %>%
.[, bmi := as.numeric(ifelse(bmi == "N/A", NA_character_, bmi))] %>%
.[, id := as.character(id)] %>%
.[gender != "Other",]
# id muss ein character sein, sonst bekomme ich unten bei der rollenzuteilung einen error
# denn ein "name" spalte muss character oder factor sein, aber nie integer - whyever
#### TASK ######################################################################
# es ist etwas kompliziert eine variable den feature status zu entziehen.
task_stroke = as_task_classif(d, target = "stroke")
task_stroke$col_roles$feature <- setdiff(task_stroke$col_roles$feature, "id")
task_stroke$col_roles$name <- "id"
task_stroke$set_col_roles("stroke", c("target","stratum"))
split = partition(task_stroke, ratio = 0.8)
### learner --------------------------------------------------------------------
learner_rf = lrn("classif.ranger",
predict_type = "prob",
respect.unordered.factors = "partition",
num.trees = 3000,
id = "rf") # mit id = "rf" kann ich bei ps dann die präambel kuerzer schreiben
learner_nb = lrn("classif.naive_bayes",
predict_type = "prob",
id = "nb")
learner_knn = lrn("classif.kknn",
predict_type = "prob",
id = "knn")
# rf muss davor geschrieben werden, weil es sonst bei der pipe unklarheiten
# bezueglich parameternamen gibt!
param_set = ps(
gabelung.selection = p_fct(levels = c("unknown_as_category", "impute_unknown")),
cvnb.laplace = p_dbl(lower = 0, upper = 2),
cvnb.eps = p_dbl(lower = 1e-6, upper = 1e-2),
cvrf.mtry.ratio = p_dbl(0.4, 1),
cvrf.min.node.size = p_int(50, 1000),
super.eta = p_dbl(lower = 0.01, upper = 0.4), # Lernrate von XGBoost
super.max_depth = p_int(lower = 3, upper = 11), # Maximale Tiefe
super.nrounds = p_int(lower = 50, upper = 1000), # Anzahl der Boosting-Runden
cvknn.k = p_int(lower = 3, upper = 60)
)
### resampling -----------------------------------------------------------------
resampling_CV5 = rsmp("cv", folds = 5)
measure_AUC = msr("classif.auc")
### tuner ----------------------------------------------------------------------
tuner_bayes = tnr("mbo")
### terminator -----------------------------------------------------------------
terminator2 = trm("combo",
list(
trm("evals", n_evals = n_evals),
trm("run_time", secs = duration)
)
)
################################################################################
### pipeline -------------------------------------------------------------------
################################################################################
gabelung = po("branch",
options = c("unknown_as_category", "impute_unknown"), id = "gabelung")
robustifiy = pipeline_robustify(task = task_stroke,
learner = learner_rf,
character_action = "factor!",
impute_missings = FALSE)
#### keep unknown ==============================================================
po_impute_f_uk <- po("imputelearner", learner = lrn("classif.rpart"),
param_vals = list(
affect_columns = selector_type("factor")
), id = "imp_f_uk") # id notwendig weil sonst probleme bei robustify
po_impute_d_uk <- po("imputelearner", learner = lrn("regr.rpart"),
param_vals = list(
affect_columns = selector_type("numeric")
), id = "imp_d_uk")
graph_impute_unknown = po("colapply",
param_vals = list(
applicator = function(x) {
# x ist hier ein Vektor der Spalte
x1 <- data.table::copy(x)
x1[x == "Unknown"] <- NA_integer_
res = forcats::fct_drop(x1)
return(res)
},
affect_columns = selector_grep("smoking_status")
), id = "unknown_as_na"
) %>>%
po_impute_f_uk %>>%
po_impute_d_uk
#### impute unknown ============================================================
po_impute_d_imp <- po("imputelearner", learner = lrn("regr.rpart"),
param_vals = list(
affect_columns = selector_type("numeric")
), id = "imp_d")
#############
super_learner = lrn("classif.rpart", predict_type = "prob", id = "super")
### stacking
base_learners =
gunion(list(
po("nop", id = "nop_original"),
po("learner_cv", learner = learner_rf, id = "cvrf"),
po("learner_cv", learner = learner_nb, id = "cvnb"),
po("learner_cv", learner = learner_knn, id = "cvknn")
)) %>>% po("featureunion", id = "base_learners")
#### COMBINED GRAPH ============================================================
combined_graph = robustifiy %>>%
gabelung %>>%
gunion(list(po_impute_d_imp, graph_impute_unknown)) %>>%
po("unbranch", id = "unbranch_main") %>>%
base_learners %>>%
po("encode", method = "one-hot", id = "encode_super") %>>%
po("learner", learner = lrn("classif.xgboost", predict_type = "prob", id = "super"))
################################################################################
### autotune -------------------------------------------------------------------
at = AutoTuner$new(
learner = combined_graph,
resampling = resampling_CV5,
measure = measure_AUC,
search_space = param_set,
terminator = terminator2,
tuner = tuner_bayes
)
at$train(task_stroke, row_ids = split$train)
pred_res = at$predict(task_stroke, row_ids = split$test)
auc_test = measure_AUC$score(pred_res)
full_res = list(at = at,
pred_res = pred_res,
auc_test = auc_test,
split = split)
saveRDS(full_res, "output/at_stack2_splitted.rds") Was können Pipelines?
Die Einzelteile einer Pipeline sind wie Mini-Modelle. Also, man kann sie auch trainieren und wir bekommen ein etwas unübersichtliches, aber komplexes Objekt zurück. Wir nehmen jetzt mal den gekürzten Anfang unseres gestackten Modells, um uns dann mit den Pipes herumspielen zu können2.
Robustify

Wenn Modelle anfangen, Errors zu droppen, zieht es mich plötzlich ganz weit weg vom Computer.
Diese vorgefertigte Pipeline, sorgt dafür, dass es zu keinen bösen Überraschungen kommt! Aus meiner Sicht: Uneingeschränkte Empfehlung. Man erspart sich viele Fehlermeldungen! Diesen Text zu lesen ist btw. auch sehr empfehlenswert! Es werden die Daten nur gerade so viel verändert, wie es für den learner unbedingt notwendig ist. Minimalistisch und zielführend.
- “factor!” erzwingt, dass
characterSpalten zufactorumgewandelt werden. Die Pipeline würde das nicht machen, wenn es für denlearnernicht notwendig wäre. - Es werden keine Missings imputiert. Das will ich dann später selbst machen.
Wir sehen hier einen Vorher-/Nachher-Vergleich. Zuerst sind noch einige character-Spalten enthalten, nachher, keine einzige mehr! Wir sehen auch, dass die Spalten umsortiert wurden. Nun sind alle factor-Spalten in einem Block.
# Vorher:
sapply(task_stroke$data(), class) stroke Residence_type age avg_glucose_level
"factor" "character" "numeric" "numeric"
bmi ever_married gender heart_disease
"numeric" "character" "character" "integer"
hypertension smoking_status work_type
"integer" "character" "character" # Anwenden der Robustify Pipe auf den Task
res_rob = robustifiy$train(task_stroke)
# Nachher:
sapply(res_rob[[1]]$data(), class) stroke Residence_type ever_married gender
"factor" "factor" "factor" "factor"
smoking_status work_type age avg_glucose_level
"factor" "factor" "numeric" "numeric"
bmi heart_disease hypertension
"numeric" "integer" "integer" Mit der plot Methode lässt sich die Pipeline samt ihrer Elemente sogar interaktiv darstellen wie in Abbildung 2 zu sehen ist!
Robustify - Mini Beispiel
Konstanten machen Probleme, wenn man sie einfach so einer statistischen Methode zuführt. Die Methode erwartet Variablen. Eine Konstante hat diesen Namen nicht verdient, denn sie variiert ja per Definition nicht. Typischerweise will man Konstanten loswerden, da sie nichts – aber auch gar nichts – Konstruktives beitragen, um die Varianz von y zu erklären. Jetzt wäre es ein leichtes, alle Konstanten zu eliminieren. Allerdings kann eine Variable, die im Gesamtdatensatz noch ausreichend Variation zeigt, durch die Gruppenbildung der Kreuzvalidierung zu einer Konstanten werden. Die Alarmglocken schrillen – die Pipe kommt zur Hilfe. Schauen wir also mal, was passiert, wenn man eine Konstante im Datensatz hat.
Hier wird aber gleich mehrerlei ausprobiert, in dem kleinen Datensatz dat_mini. Es wird gleich zweimal pipeline_robustify mit unterschiedlichen Einstellungen ausgeführt.
- Hier wird erwähnt, dass explizit nicht imputiert werden soll
- Es soll imputiert werden und es sollen alle
characterSpaltenfactorwerden, egal, ob das notwendig ist oder nicht.
# Simulation eines Mini Datensatzes
set.seed(1653)
dat_mini <- data.frame(
target = factor(sample(c("A", "B", "C"), 20, replace = TRUE)),
feature1 = rnorm(20),
feature2 = sample(letters, 20, replace = TRUE),
constant_feature = 1 # <- konstante Spalte
) %>%
data.table
dat_mini[4:5, feature1 := NA_real_] # Missings erzeugen
# Task erstellen
task_mini = as_task_classif(dat_mini, target = "target")
# Robustify
robbi_mini1 = pipeline_robustify(task = task_mini,
learner = learner_rf,
impute_missings = FALSE
)
robbi_mini2 = pipeline_robustify(task = task_mini,
learner = learner_rf,
character_action = "factor!",
impute_missings = TRUE)
# Graph trainieren
robbi_res1 = robbi_mini1$train(task_mini)
robbi_res2 = robbi_mini2$train(task_mini)Das wirkt sich direkt auf die Daten aus.
- Hier sind noch die 2 Missings enthalten und
feature2ist nochcharacteraber die Konstante ist weg! - Hier ist ebenso die Konstante weg, Missings sind weg,
charactergeändert und eine neue Spalte hier, die anzeigt, wo ursprünglich Missings waren!
target feature1 feature2
<fctr> <num> <char>
1: B -0.7315033 b
2: B 1.9773381 h
3: C 0.3296053 k
4: A NA j
5: B NA s
6: A -0.9334918 q target feature2 feature1 missing_feature1
<fctr> <fctr> <num> <fctr>
1: B b -0.7315033 present
2: B h 1.9773381 present
3: C k 0.3296053 present
4: A j 1.5194536 missing
5: B s -1.1473332 missing
6: A q -0.9334918 presentImputation

Will man sich mit fehlenden Werten nicht zufriedengeben oder verwendet man obskure statistische Methoden, die mit fehlenden Werten nicht umgehen können, ist es Zeit, sich etwas auszudenken. Der erste Impuls ist vielleicht, sofort zu imputieren, denn die fehlenden Werte sind wie die von Datenkaries gefressenen Löcher in unserem, hoffentlich ansonsten gesunden, Datenzahn. Die sofortige Imputation ist aber laut Beipackzettel nicht empfohlen und wird in Fachkreisen auch “Imputatio Praecox” genannt. Denn wer es sich leisten kann, und das sollte wirklich jeder, imputiert tunlichst innerhalb der CV Samples, mit dem Ziel, keinerlei Informationen vom CV-Trainingssample an das CV-Testsample zu liefern. Wer Style hat, imputiert also so spät wie möglich.
Der Pipeline Operator po wird wie ein Modell trainiert. Interessant ist auch, dass in dem Fall für alle numerischen Variablen ein Modell gemacht wird, egal ob es wirklich benötigt wird oder nicht. In unserem Fall gibt es also Modelle für age, avg_glucose_level und bmi. Wirklich angewendet würde es in der pipe nur für bmi. Dort dann automatisch. Wir müssen die predict Methode verwenden, um die Werte wirklich zu imputieren.
po_impute_d_imp <- po("imputelearner", learner = lrn("regr.rpart"),
param_vals = list(
affect_columns = selector_type("numeric")
), id = "imp_d")
po_impute_d_imp$train(res_rob)
tsk_imputed = po_impute_d_imp$predict(res_rob)state_imputer = po_impute_d_imp$state
trained_learners = state_imputer$model
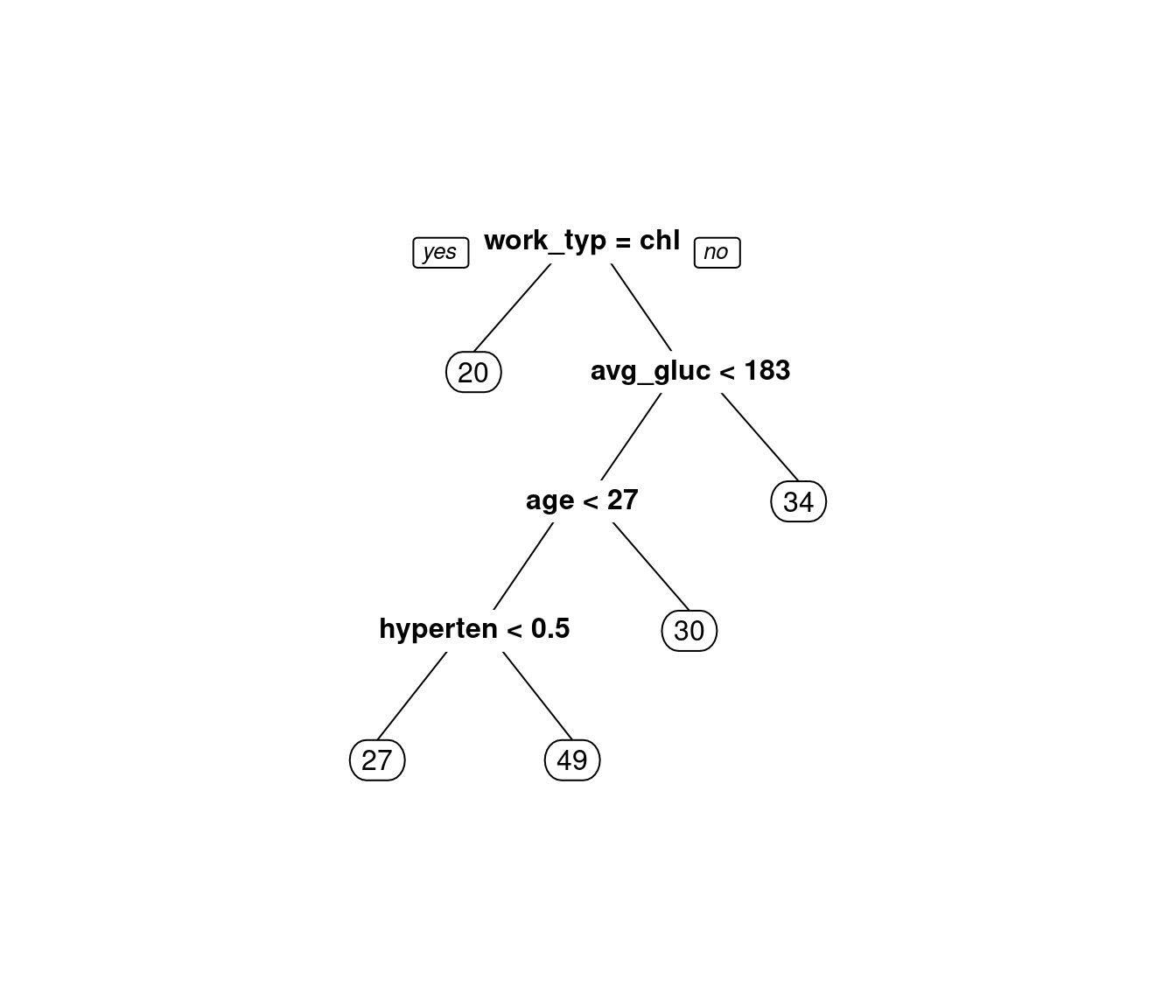
names(trained_learners)[1] "age" "avg_glucose_level" "bmi" Wir holen uns das Modell aus dem Objekt raus und können den Regression Tree, der als Methode gewählt wurde, plotten. Abbildung 3 gibt Gelegenheit dazu, die Splits, die der Baum gewählt hat, zu inspizieren. Leider dienen hier nur gerundete Werte der numerischen Einordnung. Die Autor:innen dieser schönen Funktion haben sich sehr viel Mühe gemacht, Argumente zu ersinnen, die eine mögliche Änderung des Rundungsverhaltens insinnuieren, den Zahlendruck in der Grafik aber unangetastet lassen. Mithilfe dieser Argumente lassen sich Rundungen der verschiedensten Boxen feinabstimmen, um das, dem Eckigen abgeneigte, Auge nicht zu kränken. Ob dieser irreführenden Sackgassen, sackte ich in Resignation zusammen3.
model_bmi = trained_learners[["bmi"]]$model
prp(model_bmi)Wir können auch nachvollziehen, ob und was imputiert wurde. Statt vieler Missings, sind nun Werte zu sehen, die wir dank der Rundungen in der vorigen Abbildung nicht wiedererkennen.
index_bmi_na = task_stroke$data()[,which(is.na(bmi))]
cbind(tsk_imputed$output$data(rows = index_bmi_na, cols = "bmi") %>% head,
task_stroke$data(rows = index_bmi_na, cols = "bmi") %>% head) bmi bmi
<num> <num>
1: 33.81869 NA
2: 30.34060 NA
3: 33.81869 NA
4: 33.81869 NA
5: 33.81869 NA
6: 33.81869 NAOk, Pipelines können noch viel mehr und es gäbe noch einiges herzuzeigen, aber das kommt vielleicht in einem dritten Teil. Vielleicht auch nicht.